python简单实现gillespie模拟
By 苏剑林 | 2018-06-07 | 90563位读者 |由于专业需求,需要做主方程的随机模拟。在网上并没有找到适合的Python实现,遂自己写了一个,分享一下源码。至于gillespie算法本身就不介绍了,有需要的读者自然会懂,没需要的读者不建议去懂。
源码 #
其实基本的gillespie模拟算法很简单,也很好实现,下面就是一个参考例子:
#! -*- coding: utf-8 -*-
import numpy as np
from scipy.special import comb
class Reaction: # 封装的类,代表每一个化学反应
def __init__(self, rate=0., num_lefts=None, num_rights=None):
self.rate = rate # 反应速率
assert len(num_lefts) == len(num_rights)
self.num_lefts = np.array(num_lefts) # 反应前各个反应物的数目
self.num_rights = np.array(num_rights) # 反应后各个反应物的数目
self.num_diff = self.num_rights - self.num_lefts # 改变数
def combine(self, n, s): # 算组合数
return np.prod(comb(n, s))
def propensity(self, n): # 算反应倾向函数
return self.rate * self.combine(n, self.num_lefts)
class System: # 封装的类,代表多个化学反应构成的系统
def __init__(self, num_elements):
assert num_elements > 0
self.num_elements = num_elements # 系统内的反应物的类别数
self.reactions = [] # 反应集合
def add_reaction(self, rate=0., num_lefts=None, num_rights=None):
assert len(num_lefts) == self.num_elements
assert len(num_rights) == self.num_elements
self.reactions.append(Reaction(rate, num_lefts, num_rights))
def evolute(self, steps, inits=None): # 模拟演化
self.t = [0] # 时间轨迹,t[0]是初始时间
if inits is None:
self.n = [np.ones(self.num_elements)]
else:
self.n = [np.array(inits)] # 反应物数目,n[0]是初始数目
for i in range(steps):
A = np.array([rec.propensity(self.n[-1])
for rec in self.reactions]) # 算每个反应的倾向函数
A0 = A.sum()
A /= A0 # 归一化得到概率分布
t0 = -np.log(np.random.random())/A0 # 按概率选择下一个反应发生的间隔
self.t.append(self.t[-1] + t0)
d = np.random.choice(self.reactions, p=A) # 按概率选择其中一个反应发生
self.n.append(self.n[-1] + d.num_diff)使用 #
为了方便使用,我对反应进行了封装。现在,只需要根据反应式就可以直接进行模拟了,无须额外的编程操作。比如比较简单的基因表达模型:
$$\begin{aligned}DNA &\xrightarrow{\quad 20\quad} DNA + m\\
m &\xrightarrow{\quad 2.5\quad} m + n\\
m &\xrightarrow{\,\quad 1\,\,\quad} \phi\\
n &\xrightarrow{\,\quad 1\,\,\quad} \phi
\end{aligned}$$
这里$m,n$分别代表mRNA和蛋白质的数目,$\phi$是空,意味着降解或者“无中生有”。其中第一个反应可以简化为$\phi \xrightarrow{\quad 20\quad} m$,所以实际上是两种反应物$m,n$的四个反应式。
num_elements = 2
system = System(num_elements)
system.add_reaction(20, [0, 0], [1, 0])
system.add_reaction(2.5, [1, 0], [1, 1])
system.add_reaction(1, [1, 0], [0, 0])
system.add_reaction(1, [0, 1], [0, 0])
system.evolute(100000)然后就可以统计画图:
import matplotlib.pyplot as plt
import pandas as pd
x = system.t
y = [i[1] for i in system.n]
plt.clf()
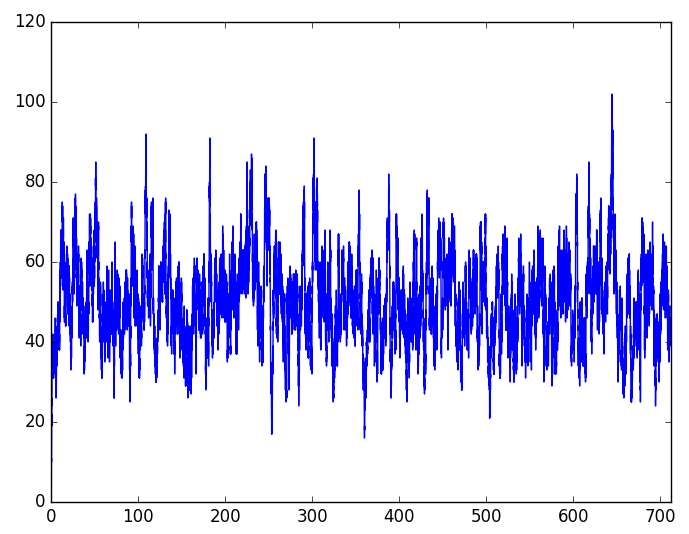
plt.plot(x, y) # 蛋白质的轨线图
plt.xlim(0, x[-1]+1)
plt.savefig('test.png')
d = pd.Series([i[1] for i in system.n]).value_counts()
d = d.sort_index()
d /= d.sum()
plt.clf()
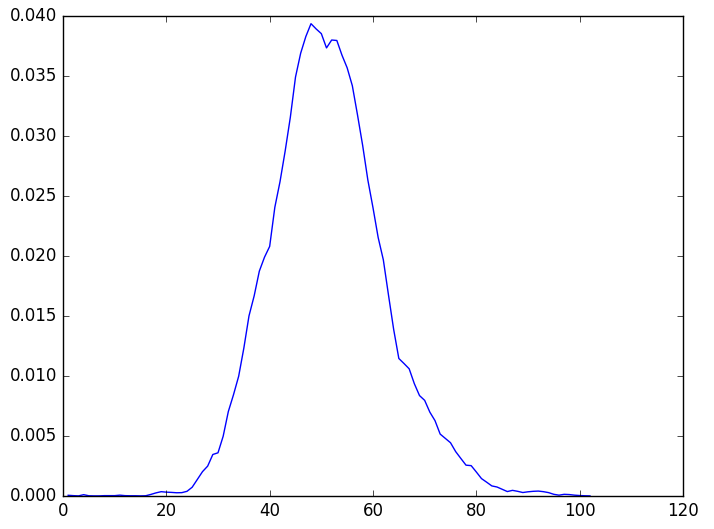
plt.plot(d.index, d) # 蛋白质的(经验)分布图
plt.savefig('test.png')结果为:
转载到请包括本文地址:https://kexue.fm/archives/5607
更详细的转载事宜请参考:《科学空间FAQ》
如果您还有什么疑惑或建议,欢迎在下方评论区继续讨论。
如果您觉得本文还不错,欢迎分享/打赏本文。打赏并非要从中获得收益,而是希望知道科学空间获得了多少读者的真心关注。当然,如果你无视它,也不会影响你的阅读。再次表示欢迎和感谢!
如果您需要引用本文,请参考:
苏剑林. (Jun. 07, 2018). 《python简单实现gillespie模拟 》[Blog post]. Retrieved from https://kexue.fm/archives/5607
@online{kexuefm-5607,
title={python简单实现gillespie模拟},
author={苏剑林},
year={2018},
month={Jun},
url={\url{https://kexue.fm/archives/5607}},
}










July 3rd, 2018
你好作者 我想问一下 你这个程序如果模拟一个复杂的反应 如:1. S+E生成SE 反应速率:0.00166 2. SE生成S+E 反应速率:0.0001 3. SE生成P+E 反应速率:0.1
初试数量:E=121 S=300 P=0 SE=0 这种反应可以用你的程序模拟吗 为何我多次尝试不行
你这个系统,有四种分子,所以
num_elements = 4
system = System(num_elements)
假设四个位置分别代表[S, E, SE, P],那么
system.add_reaction(1.66e-3, [1, 1, 0, 0], [0, 0, 1, 0])
system.add_reaction(1e-4, [0, 0, 1, 0], [1, 1, 0, 0])
system.add_reaction(0.1, [0, 0, 1, 0], [0, 1, 0, 1])
最后,system.evolute(100000, [300, 121, 0, 0])
July 9th, 2018
你好,这行ssert len(num_lefts) == self.num_elements应该有一个是assert len(num_rights) == self.num_elements吧
是的,感谢提出,已经修正。
May 14th, 2019
你好,我好像运行不出来
你好,我好像能运行出来。
June 8th, 2019
您好 请问一下最后一张“蛋白质的统计分布”图像该怎么理解呢?我不是学计算机的...实在看不懂这个...麻烦您了,十分感谢:)
就是由实验数据统计出的经验分布。不会连这个都不理解吧?你用gillespie模拟干嘛呢?
October 6th, 2019
你好作者,我想问一下,我运行的时候显示system未定义,这个是什么情况
June 23rd, 2020
你好,请问为什么我运行你的代码出来是类似折线图的图形呢
有两个图,一个是轨线图,一个是分布图,你画的是哪个?
抱歉是我自己的问题,已经调试正确了,谢谢作者的分享~
你好,我也遇到了同样的问题,请问您是怎么解决的呢?
November 26th, 2020
太难了吧
June 14th, 2021
您好 这个程序好像无法实现反应速率随反应物改变情况下的模拟,应该要再加入一个update函数更新反应式
你可以自行修改程序,以达到这种效果。
October 22nd, 2021
您好,感谢您提供的程序和解释,受益匪浅!
我不是化学专业的,关于您列出的基因表达模型有些疑问。
请问4个反应式中间箭头的上的数字表示什么?是否可以理解为这个反应通道在该反应系统中的组合数(或称为配套反应分子组合的个数)?
以及为什么第3个反应不能和第2个交换位置呢?